MEDICINA: CÓDIGO GENÉTICO. El código genético es el conjunto de normas por las que la información codificada en el material genético (secuencias de ADN o ARN) se traduce en proteínas (secuencias de aminoácidos) en las células vivas. El código define la relación entre secuencias de tres nucleótidos, llamadas codones, y aminoácidos. Un codón se corresponde con un aminoácido específico. La secuencia del material genético se compone de cuatro bases nitrogenadas distintas, que tienen una función equivalente a letras en el código genético: adenina (A), timina (T), guanina (G) y citosina (C) en el ADN y adenina (A), uracilo (U), guanina (G) y citosina (C) en el ARN. EL MATERIAL DEL CÓGIGO GENÉTICO SE COMPONE DE MINERALES. Mineral es aquella sustancia natural, homogénea, de origen inorgánico, de composición química definida (dentro de ciertos límites). Estas sustancias inorgánicas poseen una disposición ordenada de átomos de los elementos de que está compuesto, y esto da como resultado el desarrollo de superficies planas conocidas como caras. Si el mineral ha sido capaz de crecer sin interferencias, pueden generar formas geométricas características, conocidas como cristales.
Código genético
De Wikipedia, la enciclopedia libre

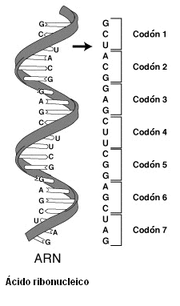
El código genético es el conjunto de normas por las que la información codificada en el material genético (secuencias de ADN o ARN) se traduce en proteínas (secuencias de aminoácidos) en las células vivas. El código define la relación entre secuencias de tres nucleótidos, llamadas codones, y aminoácidos. Un codón se corresponde con un aminoácido específico.
La secuencia del material genético se compone de cuatro bases nitrogenadas distintas, que tienen una función equivalente a letras en el código genético: adenina (A), timina (T), guanina (G) y citosina (C) en el ADN y adenina (A), uracilo (U), guanina (G) y citosina (C) en el ARN.
Debido a esto, el número de codones posibles es 64, de los cuales 61 codifican aminoácidos (siendo además uno de ellos el codón de inicio, AUG) y los tres restantes son sitios de parada (UAA, llamado ocre; UAG, llamado ámbar; UGA, llamado ópalo). La secuencia de codones determina la secuencia aminoacídica de una proteína en concreto, que tendrá una estructura y una función específicas.
Contenido[ocultar] |
Descubrimiento del código genético [editar]
Cuando James Watson, Francis Crick, Maurice Wilkins y Rosalind Franklin descubrieron la estructura del ADN, comenzó a estudiarse en profundidad el proceso de traducción en proteínas. En 1955, Severo Ochoa y Marianne Grunberg-Manago aislaron la enzima polinucleótido fosforilasa, capaz de sintetizar ARNm sin necesidad de modelo a partir de cualquier tipo de nucleótidos que hubiera en el medio. Así, a partir de un medio en el cual tan sólo hubiera UDP (urdín difosfato) se sintetizaba un ARNm en el cual únicamente se repetía el ácido urídico, el denominado poli-U (....UUUUU....). George Gamow postuló que un código de codones de tres bases debía ser el empleado por las células para codificar la secuencia aminoacídica, ya que tres es el número entero mínimo que con cuatro bases nitrogenadas distintas permiten más de 20 combinaciones (64 para ser exactos).
Los codones constan de tres nucleótidos fue demostrado por primera vez en el experimento de Crick, Brenner y colaboradores. Marshall Nirenberg y Heinrich J. Matthaei en 1961 en los Institutos Nacionales de Salud descubrieron la primera correspondencia codón-aminoácido. Empleando un sistema libre de células, tradujeron una secuencia ARN de poli-uracilo (UUU...) y descubrieron que el polipéptido que habían sintetizado sólo contenía fenilalanina. De esto se deduce que el codón UUU especifica el aminoácido fenilalanina. Continuando con el trabajo anterior, Nirenberg y Philip Leder fueron capaces de determinar la traducción de 54 codones, utilizando diversas combinaciones de ARNm, pasadas a través de un filtro que contiene ribosomas. Los ARNt se unían a tripletes específicos.
Posteriormente, Har Gobind Khorana completó el código, y poco después, Robert W. Holley determinó la estructura del ARN de transferencia, la molécula adaptadora que facilita la traducción. Este trabajo se basó en estudios anteriores de Severo Ochoa, quien recibió el premio Nobel en 1959 por su trabajo en la enzimología de la síntesis de ARN. En 1968, Khorana, Holley y Nirenberg recibieron el Premio Nobel en Fisiología o Medicina por su trabajo.
Transferencia de información [editar]
El genoma de un organismo se encuentra en el ADN o, en el caso de algunos virus, en el ARN. La porción de genoma que codifica una proteína o un ARN se conoce como gen. Esos genes que codifican proteínas están compuestos por unidades de trinucleótidos llamadas codones, cada una de los cuales codifica un aminoácido. Cada subunidad nucleotídica está formada por un fosfato, una desoxirribosa y una de las cuatro posibles bases nitrogenadas. Las bases purínicas adenina (A) y guanina (G) son más grandes y tienen dos anillos aromáticos. Las bases pirimidínicas citosina (C) y timina (T) son más pequeñas y sólo tienen un anillo aromático. En la configuración en doble hélice, dos cadenas de ADN están unidas entre sí por puentes de hidrógeno en una asociación conocida como emparejamiento de bases. Además, estos puentes siempre se forman entre una adenina de una cadena y una timina de la otra y entre una citosina de una cadena y una guanina de la otra. Esto quiere decir que el número de residuos A y T será el mismo en una doble hélice y lo mismo pasará con el número de residuos de G y C. En el ARN, la timina (T) se sustituye por uracilo (U), y la desoxirribosa por una ribosa.
Cada gen codificante de proteína se transcribe en una molécula plantilla, que se conoce como ARN mensajero o ARNm. Éste, a su vez, se traduce en el ribosoma, en una cadena aminoacídica o polipeptídica. En el proceso de traducción se necesita un ARN de transferencia específico para cada aminoácido con el aminoácido unido a él covalentemente, guanosina trifosfato como fuente de energía y ciertos factores de traducción. Los ARNt tienen anticodones complementarios a los codones del ARNm y se pueden “cargar” covalentemente en su extremo 3' terminal CCA con aminoácidos. Los ARNt individuales se cargan con aminoácidos específicos por las enzimas llamadas aminoacil ARNt sintetasas, que tienen alta especificidad tanto por aminoácidos como por ARNt. La alta especificidad de estas enzimas es motivo fundamental del mantenimiento de la fidelidad de la traducción de proteínas.
Hay 4³ = 64 combinaciones diferentes de codones que sean posibles con tripletes de tres nucleótidos: los 64 codones están asignados a aminoácido o a señales de parada en la traducción. Si, por ejemplo, tenemos una secuencia de ARN, UUUAAACCC, y la lectura del fragmento empieza en la primera U (convenio 5' a 3'), habría tres codones que serían UUU, AAA y CCC, cada uno de los cuales especifica un aminoácido. Esta secuencia de ARN se traducirá en una secuencia aminoacídica de tres aminoácidos de longitud. Se puede comparar con la informática, donde un codón se asemejaría a una palabra, lo que sería el “trozo” estándar para el manejo de datos (como un aminoácido a una proteína), y un nucleótido es similar a un bit, que sería la unidad más pequeña. (En la práctica, se necesitarían al menos 2 bits para representar un nucleótido, y 6 para un codón, en un ordenador normal).
Características [editar]
Universalidad [editar]
El código genético es compartido por todos los organismos conocidos, incluyendo virus y organulos, aunque pueden aparecer pequeñas diferencias. Así, por ejemplo, el codón UUU codifica para el animoácido fenilalanina tanto en bacterias, como en arqueas, como en eucariontes. Este hecho indica que el código genético ha tenido un origen único en todos los seres vivos conocidos.
Gracias a la genética molecular, se han distinguido 22 códigos genéticos, que se diferencian del llamado código genético estándar por el significado de uno o más codones. La mayor diversidad se presenta en las mitocondrias, orgánulos de las células eucariotas que se originaron evolutivamente a partir de miembros del dominio Bacteria a través de un proceso de endosimbiosis. El genoma nuclear de los eucariotas sólo suele diferenciarse del código estándar en los codones de iniciación y terminación.
Especificidad y continuidad [editar]
Ningún codón codifica más de un aminoácido, ya que, de no ser así, conllevaría problemas considerables para la síntesis de proteínas específicas para cada gen. Tampoco presenta solapamiento: los tripletes se hallan dispuesto de manera lineal y continua, de manera que entre ellos no existan comas ni espacios y sin compartir ninguna base nitrogenada. Su lectura se hace en un solo sentido (5' - 3'), desde el codón de iniciación hasta el codón de parada. Sin embargo, en un mismo ARNm pueden existir varios codones de inicio, lo que conduce a la síntesis de varios polipéptidos diferentes a partir del mismo transcrito.
Degeneración [editar]
El código genético tiene redundancia pero no ambigüedad (ver las tablas de codones). Por ejemplo, aunque los codones GAA y GAG especifican los dos el ácido glutámico (redundancia), ninguno especifica otro aminoácido (no ambigüedad). Los codones que codifican un aminoácido pueden diferir en alguna de sus tres posiciones, por ejemplo, el ácido glutámico se especifica por GAA y GAG (difieren en la tercera posición), el aminoácido leucina se especifica por los codones UUA, UUG, CUU, CUC, CUA y CUG (difieren en la primera o en la tercera posición), mientras que en el caso de la serina, se especifica por UCA, UCG, UCC, UCU, AGU, AGC (difieren en la primera, segunda o tercera posición).
De una posición de un codón se dice que es cuatro veces degenerada si con cualquier nucleótido en esta posición se especifica el mismo aminoácido. Por ejemplo, la tercera posición de los codones de la glicina (GGA, GGG, GGC, GGU) es cuatro veces degenerada, porque todas las sustituciones de nucleótidos en este lugar son sinónimos; es decir, no varían el aminoácido. Sólo la tercera posición de algunos codones puede ser cuatro veces degenerada. Se dice que una posición de un codón es dos veces degenerada si sólo dos de las cuatro posibles sustituciones de nucleótidos especifican el mismo aminoácido. Por ejemplo, la tercera posición de los codones del ácido glutámico (GAA, GAG) es doble degenerada. En los lugares dos veces degenerados, los nucleótidos equivalentes son siempre dos purinas (A/G) o dos pirimidinas (C/U), así que sólo sustituciones transversionales (purina a pirimidina o pirimidina a purina) en dobles degenerados son antónimas. Se dice que una posición de un codón es no degenerada si una mutación en esta posición tiene como resultado la sustitución de un aminoácido. Sólo hay un sitio triple degenerado en el que cambiando tres de cuatro nucleótidos no hay efecto en el aminoácido, mientras que cambiando los cuatro posibles nucleótidos aparece una sustitución del aminoácido. Esta es la tercera posición de un codón de isoleucina: AUU, AUC y AUA, todos codifican isoleucina, pero AUG codifica metionina. En biocomputación, este sitio se trata a menudo como doble degenerado.
Hay tres aminoácidos codificados por 6 codones diferentes: serina, leucina, arginina. Sólo dos aminoácidos se especifican por un único codón; uno de ellos es la metionina, especificado por AUG, que también indica el comienzo de la traducción; el otro es triptófano, especificado por UGG. Que el código genético sea degenerado es lo que determina la posibilidad de mutaciones sinónimas.
La degeneración aparece porque el código genético designa 20 aminoácidos y la señal parada. Debido a que hay cuatro bases, los codones en triplete se necesitan para producir al menos 21 códigos diferentes. Por ejemplo, si hubiera dos bases por codón, entonces sólo podrían codificarse 16 aminoácidos (4²=16). Y dado que al menos se necesitan 21 códigos, 4³ da 64 codones posibles, indicando que debe haber degeneración.
Esta propiedad del código genético lo hacen más tolerante a los fallos en mutaciones puntuales. Por ejemplo, en teoría, los codones cuatro veces degenerados pueden tolerar cualquier mutación puntual en la tercera posición, aunque el codón de uso sesgado restringe esto en la práctica en muchos organismos; los dos veces degenerados pueden tolerar una de las tres posibles mutaciones puntuales en la tercera posición. Debido a que las mutaciones de transición (purina a purina o pirimidina a pirimidina) son más probables que las de transversión (purina a pirimidina o viceversa), la equivalencia de purinas o de pirimidinas en los lugares dobles degenerados añade una tolerancia a los fallos complementaria.
Agrupamiento de codones por residuos aminoacídicos, volumen molar e hidropatía [editar]
Una consecuencia práctica de la redundancia es que algunos errores del código genético sólo causen una mutación silenciosa o un error que no afectará a la proteína porque la hidrofilidad o hidrofobidad se mantiene por una sustitución equivalente de aminoácidos; por ejemplo, un codón de NUN (N =cualquier nucleótido) tiende a codificar un aminoácido hidrofóbico. NCN codifica residuos aminoacídicos que son pequeños en cuanto a tamaño y moderados en cuanto a hidropatía; NAN codifica un tamaño promedio de residuos hidrofílicos; UNN codifica residuos que no son hidrofílicos.[1] [2] Estas tendencias pueden ser resultado de una relación de las aminoacil ARNt sintetasas con los codones heredada un ancestro común de los seres vivos conocidos.
Incluso así, las mutaciones puntuales pueden causar la aparición de proteínas disfuncionales. Por ejemplo, un gen de hemoglobina mutado provoca la enfermedad de células falciformes. En la hemoglobina mutante un glutamato hidrofílico (Glu) se sustituye por una valina hidrofóbica (Val), es decir, GAA o GAG se convierte en GUA o GUG. La sustitución de glutamato por valina reduce la solubilidad de β-globina que provoca que la hemoglobina forme polímeros lineales unidos por interacciones hidrofóbicas entre los grupos de valina y causando la deformación falciforme de los eritrocitos. La enfermedad de las células falciformes no está causada generalmente por una mutación de novo. Más bien se selecciona en regiones de malaria (de forma parecida a la talasemia), ya que los individuos heterocigotos presentan cierta resistencia ante el parásito malárico Plasmodium (ventaja heterocigótica o heterosis).
La relación entre el ARNm y el ARNt a nivel de la tercera base se puede producir por bases modificadas en la primera base del anticodón del ARNt, y los pares de bases formados se llaman “pares de bases wobble” (tambaleantes). Las bases modificadas incluyen inosina y los pares de bases que no son del tipo Watson-Crick U-G.
Usos incorrectos del término [editar]
La expresión "código genético" es frecuentemente utilizada en los medios de comunicación como sinónimo de genoma, de genotipo, o de ADN. Frases como «Se analizó el código genético de los restos y coincidió con el de la desaparecida», o «se creará una base de datos con el código genético de todos los ciudadanos» son científicamente incorrectas. Es insensato, por ejemplo, aludir al «código genético de una determinada persona», porque el código genético es el mismo para todos los individuos. Sin embargo, cada organismo tiene un genotipo propio, aunque es posible que lo comparta con otros si se ha originado por algún mecanismo de multiplicación asexual.
Tabla del código genético estándar [editar]
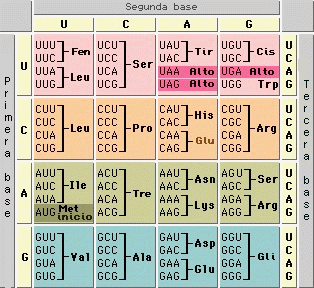
El código genético estándar se refleja en las siguientes tablas. La tabla 1 muestra qué aminoácido especifica cada uno de los 64 codones. La tabla 2 muestra qué codones especifican cada uno de los 20 aminoácidos que intervienen en la traducción. Estas tablas se llaman tablas de avance y retroceso respectivamente. Por ejemplo, el codón AAU es el aminoácido asparagina, y UGU y UGC representan cisteína (en la denominación estándar por 3 letras, Asn y Cys, respectivamente).
| apolar | polar | básico | ácido | codón de parada |
| 2ª base | |||||
|---|---|---|---|---|---|
| U | C | A | G | ||
| 1ª base | U | UUU (Phe/F) Fenilalanina UUC (Phe/F) Fenilalanina | UCU (Ser/S) Serina UCC (Ser/S) Serina | UAU (Tyr/Y) Tirosina UAC (Tyr/Y) Tirosina | UGU (Cys/C) Cisteína UGC (Cys/C) Cisteína |
| UUA (Leu/L) Leucina | UCA (Ser/S) Serina | UAA Parada (Ocre) | UGA Parada (Ópalo) | ||
| UUG (Leu/L) Leucina | UCG (Ser/S) Serina | UAG Parada (Ámbar) | UGG (Trp/W) Triptófano | ||
| C | CUU (Leu/L) Leucina CUC (Leu/L) Leucina | CCU (Pro/P) Prolina CCC (Pro/P) Prolina | CAU (His/H) Histidina CAC (His/H) Histidina | CGU (Arg/R) Arginina CGC (Arg/R) Arginina | |
| CUA (Leu/L) Leucina CUG (Leu/L) Leucina | CCA (Pro/P) Prolina CCG (Pro/P) Prolina | CAA (Gln/Q) Glutamina CAG (Gln/Q) Glutamina | CGA (Arg/R) Arginina CGG (Arg/R) Arginina | ||
| A | AUU (Ile/I) Isoleucina AUC (Ile/I) Isoleucina | ACU (Thr/T) Treonina ACC (Thr/T) Treonina | AAU (Asn/N) Asparagina AAC (Asn/N) Asparagina | AGU (Ser/S) Serina AGC (Ser/S) Serina | |
| AUA (Ile/I) Isoleucina | ACA (Thr/T) Treonina | AAA (Lys/K) Lisina | AGA (Arg/R) Arginina | ||
| AUG (Met/M) Metionina, Comienzo | ACG (Thr/T) Treonina | AAG (Lys/K) Lisina | AGG (Arg/R) Arginina | ||
| G | GUU (Val/V) Valina GUC (Val/V) Valina | GCU (Ala/A) Alanina GCC (Ala/A) Alanina | GAU (Asp/D) Ácido aspártico GAC (Asp/D) Ácido aspártico | GGU (Gly/G) Glicina GGC (Gly/G) Glicina | |
| GUA (Val/V) Valina GUG (Val/V) Valina | GCA (Ala/A) Alanina GCG (Ala/A) Alanina | GAA (Glu/E) Ácido glutámico GAG (Glu/E) Ácido glutámico | GGA (Gly/G) Glicina GGG (Gly/G) Glicina | ||
Nótese que el codón AUG codifica para la metionina pero además sirve de sitio de iniciación; el primer AUG en un ARNm es la región que codifica el sitio donde la traducción de proteínas se inicia.
La siguiente tabla inversa indica qué codones codifican cada uno de los aminoácidos.
| Ala (A) | GCU, GCC, GCA, GCG | Lys (K) | AAA, AAG |
|---|---|---|---|
| Arg (R) | CGU, CGC, CGA, CGG, AGA, AGG | Met (M) | AUG |
| Asn (N) | AAU, AAC | Phe (F) | UUU, UUC |
| Asp (D) | GAU, GAC | Pro (P) | CCU, CCC, CCA, CCG |
| Cys (C) | UGU, UGC | Sec (U) | UGA |
| Gln (Q) | CAA, CAG | Ser (S) | UCU, UCC, UCA, UCG, AGU, AGC |
| Glu (E) | GAA, GAG | Thr (T) | ACU, ACC, ACA, ACG |
| Gly (G) | GGU, GGC, GGA, GGG | Trp (W) | UGG |
| His (H) | CAU, CAC | Tyr (Y) | UAU, UAC |
| Ile (I) | AUU, AUC, AUA | Val (V) | GUU, GUC, GUA, GUG |
| Leu (L) | UUA, UUG, CUU, CUC, CUA, CUG | ||
| Comienzo | AUG | Parada | UAG, UGA, UAA |
Aminoácidos 21 y 22 [editar]
Exiten otros dos aminoácidos codificados por el código genético en algunas circunstancias y en algunos organismos. Son la seleniocisteína y la pirrolisina.
La selenocisteína (Sec/U)[3] es un aminoácido presente en multitud de enzimas (glutatión peroxidasas, tetraiodotironina 5' deiodinasas, tioredoxina reductasas, formiato deshidrogenasas, glicina reductasas y algunas hidrogenasas). Está codificado por el codón UGA (que normalmente es de parada) cuando están presentes en la secuencia los elementos SECIS (Secuencia de inserción de la seleniocisteína).
El otro aminoácido, la pirrolisina (Pyl/O),[4] [5] es un aminoácido presente en enzimas metabólicas de arqueas metanógenas. Está codificado por el codón UAG (que normalmente es de parada) cuando están presentes en la secuencia los elementos PYLIS (Secuencia de inserción de la pirrolisina).
Excepciones a la universalidad [editar]
Como se mencionó con anterioridad, se conocen 22 códigos genéticos. He aquí algunas diferencias con el estándar:
| Mitocondrias de vertebrados | AGA | Ter | * |
| AGG | Ter | * | |
| AUA | Met | M | |
| UGA | Trp | W | |
| Mitocondrias de invertebrados | AGA | Ser | S |
| AGG | Ser | S | |
| AUA | Met | M | |
| UGA | Trp | W | |
| AGG | Ausente en Drosophila | ||
| Mitocondrias de levaduras | AUA | Met | M |
| CUU | Thr | T | |
| CUC | Thr | T | |
| CUA | Thr | T | |
| CUG | Thr | T | |
| UGA | Trp | W | |
| CGA | Ausente | ||
| CGC | Ausente | ||
| Ciliados, Dasycladaceae y Hexamita (núcleo) | UAA | Gln | Q |
| UAG | Gln | Q | |
| Mitocondrias de mohos, protozoos y Coelenterate Mycoplasma y Spiroplasma (núcleo) | UGA | Trp | W |
| Mitocondrias de equinodermos y platelmintos | AAA | Asn | N |
| AGA | Ser | S | |
| AGG | Ser | S | |
| UGA | Trp | W | |
| Euplotidae (núcleo) | UGA | Cys | C |
| Endomycetales (núcleo) | CUG | Ser | S |
| Mitocondrias de Ascidiacea | AGA | Gly | G |
| AGG | Gly | G | |
| AUA | Met | M | |
| UGA | Trp | W | |
| Mitocondrias de platelmintos (alternativo) | AAA | Asn | N |
| AGA | Ser | S | |
| AGG | Ser | S | |
| UAA | Tyr | Y | |
| UGA | W | ||
| Blepharisma (núcleo) | UAG | Gln | Q |
| Mitocondrias de Chlorophyceae | TAG | Leu | L |
| Mitocondrias de trematodos | TGA | Trp | W |
| ATA | Met | M | |
| AGA | Ser | S | |
| AGG | Ser | S | |
| AAA | Asn | N | |
| Mitocondrias de Scenedesmus obliquus | TCA | Ter | * |
| TAG | Leu | L | |
El origen del código genético [editar]
A pesar de las variaciones que existen, los códigos genéticos utilizados por todas las formas conocidas de vida son muy similares. Esto sugiere que el código genético se estableció muy temprano en la historia de la vida y que tiene un origen común en las formas de vida actuales. Análisis filogenético sugiere que las moléculas ARNt evolucionaron antes que el actual conjunto de aminoacil-ARNt sintetasas[6] .
El código genético no es una asignación aleatoria de los codones a aminoácidos.[7] Por ejemplo, los aminoácidos que comparten la misma vía biosintética tienden a tener la primera base igual en sus codones[8] y aminoácidos con propiedades físicas similares tienden a tener similares a codones.[9] [10]
Experimentos recientes demuestran que algunos aminoácidos tienen afinidad química selectiva por sus codones.[11] Esto sugiere que el complejo mecanismo actual de traducción del ARNm que implica la acción ARNt y enzimas asociadas, puede ser un desarrollo posterior y que, en un principio, las proteínas se sintetizaran directamente sobre la secuencia de ARN, actuando éste como ribozima y catalizando la formación de enlaces peptídicos (tal como ocurre con el ARNr 23S del ribosoma).
Se ha planteado la hipótesis de que el código genético estándar actual surgiera por expansión biosintética de un código simple anterior. La vida primordial pudo adicionar nuevos aminoácidos (por ejemplo, subproductos del metabolismo), algunos de los cuales se incorporaron más tarde a la maquinaria de codificación genética. Se tienen pruebas, aunque circunstanciales, de que formas de vida primitivas empleaban un menor número de aminoácidos diferentes,[12] aunque no se sabe con exactitud que aminoácidos y en que orden entraron en el código genético.
Otro factor interesante a tener en cuenta es que la selección natural ha favorecido la degeneración del código para minimizar los efectos de las mutaciones y es debido a la interacion de dos atomos distintos en la reaccion[13] . Esto ha llevado a pensar que el código genético primitivo podría haber constado de codones de dos nucleótidos, lo que resulta bastante coherente con la hipótesis del balanceo del ARNt durante su acoplamiento (la tercera base no establece puentes de hidrógeno de Watson y Crick).
Referencias [editar]
- ↑ Yang et al. 1990. In Reaction Centers of Photosynthetic Bacteria. M.-E. Michel-Beyerle. (Ed.) (Springer-Verlag, Germany) 209-218
- ↑ Genetic Algorithms and Recursive Ensemble Mutagenesis in Protein Engineering http://www.complexity.org.au/ci/vol01/fullen01/html/
- ↑ IUPAC-IUBMB Joint Commission on Biochemical Nomenclature (JCBN) and Nomenclature Committee of IUBMB (NC-IUBMB) (1999). «Newsletter 1999». European Journal of Biochemistry 264 (2): pp. 607–609. doi:. http://www.chem.qmul.ac.uk/iubmb/newsletter/1999/item3.html.
- ↑ John F. Atkins and Ray Gesteland (2002).. The 22nd Amino Acid. Science 296 (5572): 1409–1410..
- ↑ Krzycki J (2005).. The direct genetic encoding of pyrrolysine. Curr Opin Microbiol 8 (6): 706–712..
- ↑ De Pouplana, L.R.; Turner, R.J.; Steer, B.A.; Schimmel, P. (1998). «Genetic code origins: tRNAs older than their synthetases?». Proceedings of the National Academy of Sciences 95 (19): pp. 11295. doi:. PMID 9736730. http://www.pnas.org/cgi/content/full/95/19/11295.
- ↑ Freeland SJ, Hurst LD (September 1998). «The genetic code is one in a million». J. Mol. Evol. 47 (3): pp. 238–48. doi:. PMID 9732450. http://link.springer-ny.com/link/service/journals/00239/bibs/47n3p238.html.
- ↑ Taylor FJ, Coates D (1989). «The code within the codons». BioSystems 22 (3): pp. 177–87. doi:. PMID 2650752.
- ↑ Di Giulio M (October 1989). «The extension reached by the minimization of the polarity distances during the evolution of the genetic code». J. Mol. Evol. 29 (4): pp. 288–93. doi:. PMID 2514270.
- ↑ Wong JT (February 1980). «Role of minimization of chemical distances between amino acids in the evolution of the genetic code». Proc. Natl. Acad. Sci. U.S.A. 77 (2): pp. 1083–6. doi:. PMID 6928661.
- ↑ Knight, R.D. and Landweber, L.F. (1998). Rhyme or reason: RNA-arginine interactions and the genetic code. Chemistry & Biology 5(9), R215-R220. PDF version of manuscript
- ↑ Brooks, Dawn J.; Fresco, Jacques R.; Lesk, Arthur M.; and Singh, Mona. (2002). Evolution of Amino Acid Frequencies in Proteins Over Deep Time: Inferred Order of Introduction of Amino Acids into the Genetic Code. Molecular Biology and Evolution 19, 1645-1655.
- ↑ Freeland S.J.; Wu T. and Keulmann N. (2003) The Case for an Error Minimizing Genetic Code. Orig Life Evol Biosph. 33(4-5), 457-77.
Véase también [editar]
Enlaces externos [editar]
Servicios online para convertir DNA en proteína
- DNA to Amino Acid Conversion
- DNA Sequence -> Protein Sequence converter
- DNA to protein translation (6 frames/17 genetic codes)
Tablas del código genético
Revisiones
- Niremberg y Khorana, los hackers del ADN, en el Museo Virtual Interactivo sobre la Genética y el ADN.
0 comentarios