PENSAMIENTO MÁGICO6: METABOLISMO Y PENSAMIENTO MÁGICO. ESTRUCTURAS, ESTÍMULOS, SENTIMIENTOS, PROCESOS FÍSICO-QUÍMICOS, BIOQUÍMICA (ALIMENTACIÓN MODERNA), CONTACTO CON LA CÉLULA Y REACCIÓN AL ESTÍMULO. El metabolismo es el conjunto de reacciones bioquímicas y procesos físico-químicos que ocurren en una célula y en el organismo.[1] Estos complejos procesos interrelacionados son la base de la vida a escala molecular, y permiten las diversas actividades de las células: crecer, reproducirse, mantener sus estructuras, responder a estímulos, etc.
Metabolismo
De Wikipedia, la enciclopedia libre
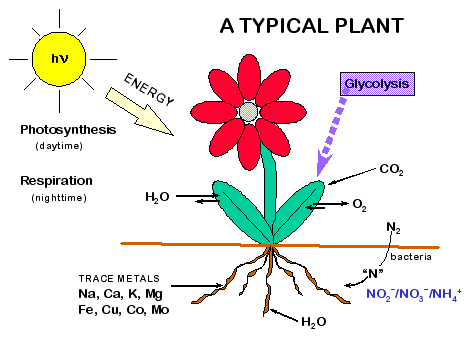

El metabolismo es el conjunto de reacciones bioquímicas y procesos físico-químicos que ocurren en una célula y en el organismo.[1] Estos complejos procesos interrelacionados son la base de la vida a escala molecular, y permiten las diversas actividades de las células: crecer, reproducirse, mantener sus estructuras, responder a estímulos, etc.
El metabolismo se divide en dos procesos conjugados: catabolismo y anabolismo. Las reacciones catabólicas liberan energía; un ejemplo es la glucólisis, un proceso de degradación de compuestos como la glucosa, cuya reacción resulta en la liberación de la energía retenida en sus enlaces químicos. Las reacciones anabólicas, en cambio, utilizan esta energía liberada para recomponer enlaces químicos y construir componentes de las células como lo son las proteínas y los ácidos nucleicos. El catabolismo y el anabolismo son procesos acoplados que hacen al metabolismo en conjunto, puesto que cada uno depende del otro.
La economía que la actividad celular impone sobre sus recursos obliga a organizar estrictamente las reacciones químicas del metabolismo en vías o rutas metabólicas, donde un compuesto químico (sustrato) es transformado en otro (producto), y este a su vez funciona como sustrato para generar otro producto, siguiendo una secuencia de reacciones bajo la intervención de diferentes enzimas (generalmente una para cada sustrato-reacción). Las enzimas son cruciales en el metabolismo porque agilizan las reacciones físico-químicas, pues hacen que posibles reacciones termodinámicas deseadas pero "desfavorables", mediante un acoplamiento, resulten en reacciones favorables. Las enzimas también se comportan como factores reguladores de las vías metabólicas, modificando su funcionalidad –y por ende, la actividad completa de la vía metabólica– en respuesta al ambiente y necesidades de la célula, o según señales de otras células.
El metabolismo de un organismo determina qué sustancias encontrará nutritivas y cuáles encontrará tóxicas. Por ejemplo, algunas procariotas utilizan sulfuro de hidrógeno como nutriente, pero este gas es venenoso para los animales.[2] La velocidad del metabolismo, el rango metabólico, también influye en cuánto alimento va a requerir un organismo.
Una característica del metabolismo es la similitud de las rutas metabólicas básicas incluso entre especies muy diferentes. Por ejemplo: la secuencia de pasos químicos en una vía metabólica como el ciclo de Krebs es universal entre células vivientes tan diversas como la bacteria unicelular Escherichia coli y organismos pluricelulares como el elefante.[3] Esta estructura metabólica compartida es muy probablemente el resultado de la alta eficiencia de estas rutas, y de su temprana aparición en la historia evolutiva.[4] [5]
Contenido[ocultar] |
Investigación y manipulación [editar]
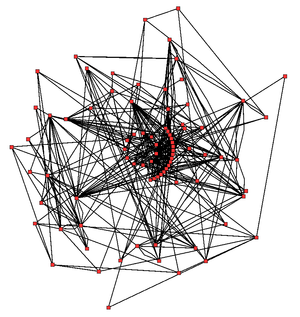
Clásicamente, el metabolismo se estudia por una aproximación reduccionista que se concentra en una ruta metabólica específica. La utilización de los diversos elementos en el organismo son valiosos en todas las categorías histológicas, de tejidos a células, que definen las rutas de precursores hacia su producto final.[6] Las enzimas que catabolizan estas reacciones químicas pueden ser purificadas y así estudiar su cinética enzimática y las respuestas que presentan frente a diversos inhibidores. Otro tipo de estudio que se puede llevar a cabo en paralelo es la identificación de los metabolitos presentes en una célula o tejido; al estudio de todo el conjunto de estas moléculas se le denomina metabolómica. Estos estudios ofrecen una visión de las estructuras y funciones de rutas metabólicas simples, pero son inadecuados cuando se quieren aplicar a sistemas más complejos como el metabolismo global de la célula.[7]
En la imagen de la derecha se puede apreciar la complejidad de una red metabólica celular que muestra interacciones entre tan sólo 43 proteínas y 40 metabolitos: esta secuencia de genomas provee listas que contienen hasta 45.000 genes.[8] Sin embargo, es posible usar esta información para reconstruir redes completas de comportamientos bioquímicos y producir más modelos matemáticos holísticos que puedan explicar y predecir su comportamiento.[9] Estos modelos son mucho más efectivos cuando se usan para integrar la información obtenida de las rutas y de los metabolitos mediante métodos clásicos con los datos de expresión génica obtenidos mediante estudios de proteómica y de chips de ADN.[10]
Una de las aplicaciones tecnológicas de esta información es la ingeniería metabólica. Con esta tecnología, organismos como las levaduras, las plantas o las bacterias son modificados genéticamente para hacerlos más útiles en algún campo de la biotecnología, como puede ser la producción de drogas, antibióticos o químicos industriales.[11] [12] [13] Estas modificaciones genéticas tienen como objetivo reducir la cantidad de energía usada para producir el producto, incrementar los beneficios y reducir la producción de desechos.[14]
Biomoléculas principales [editar]
La mayor parte de las estructuras que componen a los animales, plantas y microbios pertenecen a alguno de estos tres tipos de moléculas básicas: aminoácidos, glúcidos y lípidos (también denominados grasas). Como estas moléculas son vitales para la vida, el metabolismo se centra en sintetizar estas moléculas, en la construcción de células y tejidos, o en degradarlas y utilizarlas como recurso energético en la digestión. Muchas biomoléculas pueden interaccionar entre sí para crear polímeros como el ADN (ácido desoxirribonucleico) y las proteínas. Estas macromoléculas son esenciales en los organismos vivos. En la siguiente tabla se muestran los biopolímeros más comunes:
| Tipo de molécula | Nombre de forma de monómero | Nombre de formas de polímero |
|---|---|---|
| Péptidos | Aminoácidos | Proteínas (o polipéptidos) |
| Carbohidratos | Monosacáridos | Polisacáridos |
| Ácidos nucleicos | Nucleótidos | Polinucleótidos |
Aminoácidos y proteínas [editar]
Las proteínas están compuestas por los aminoácidos, dispuestos en una cadena lineal y unidos por enlaces peptídicos. Las enzimas son proteínas que catalizan las reacciones químicas en el metabolismo. Otras proteínas tienen funciones estructurales o mecánicas, como las proteínas del citoesqueleto que forman un sistema de andamiaje para mantener la forma de la célula.[15] [16] Las proteínas también son partícipes de la comunicación celular, la respuesta inmune, la adhesión celular y el ciclo celular.[17]
Lípidos [editar]
Los lípidos son las biomoléculas que más diversidad presentan. Su función estructural básica es formar parte de las membranas biológicas como la membrana celular, o bien como recurso energético.[17] Los lípidos son definidos normalmente como moléculas hidrófobicas o anfipáticas, que se disuelven en solventes orgánicos como la bencina o el cloroformo.[18] Las grasas son un grupo de compuestos que incluyen ácidos grasos y glicerol; una molécula de glicerol junto a tres ácidos grasos éster dan lugar a una molécula de triglicérido.[19] Se pueden dar variaciones de esta estructura básica, que incluyen cadenas laterales como la esfingosina de los esfingolípidos y los grupos hidrofílicos tales como los grupos fosfato en los fosfolípidos. Esteroides como el colesterol son otra clase mayor de lípidos sintetizados en las células.[20]
Carbohidratos [editar]
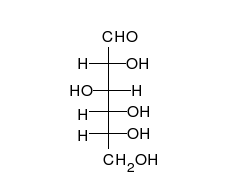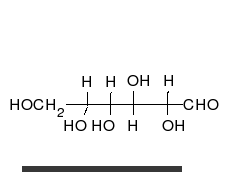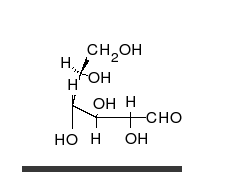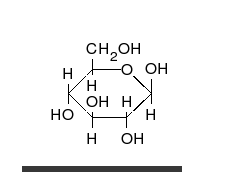
Los carbohidratos son aldehídos o cetonas con grupos hidroxilo que pueden existir como cadenas o anillos. Los carbohidratos son las moléculas biológicas más abundantes, y presentan varios papeles en la célula; algunos actúan como moléculas de almacenamiento de energía (almidón y glucógeno) o como componentes estructurales (celulosa en las plantas, quitina en los animales).[17] Los carbohidratos básicos son llamados monosacáridos e incluyen galactosa, fructosa, y el más importante la glucosa. Los monosacáridos pueden sintetizarse y formar polisacáridos.[21]
Nucleótidos [editar]
Los polímeros de ADN (ácido desoxirribonucléico) y ARN (ácido ribonucléico) son cadenas de nucleótidos. Estas moléculas son críticas para el almacenamiento y uso de la información genética por el proceso de transcripción y biosíntesis de proteínas.[17] Esta información se encuentra protegida por un mecanismo de reparación del ADN y duplicada por un mecanismo de replicación del ADN. Algunos virus tienen un genoma de ARN, por ejemplo el HIV, y utilizan retrotranscripción para crear ADN a partir de su genoma viral de ARN;[22] estos virus son denominados retrovirus. El ARN de ribozimas como los ribosomas es similar a las enzimas y puede catabolizar reacciones químicas. Los nucleósidos individuales son sintentizados mediante la unión de bases nitrogenadas con ribosa. Estas bases son anillos heterocíclicos que contienen nitrógeno y, según presenten un anillo o dos, pueden ser clasificadas como pirimidinas o purinas, respectivamente. Los nucleótidos también actúan como coenzimas en reacciones metabólicas de transferencia en grupo.[23]
Coenzimas [editar]
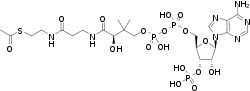
El metabolismo conlleva un gran número de reacciones químicas, pero la gran mayoría presenta alguno de los mecanismos de catálisis básicos de reacción de transferencia en grupo.[24] Esta química común permite a las células utilizar una pequeña colección de intermediarios metabólicos para trasladar grupos químicos funcionales entre diferentes reacciones.[23] Estos intermediarios de transferencia de grupos son denominados coenzimas. Cada clase de reacción de grupo es llevada a cabo por una coenzima en particular, que es el sustrato para un grupo de enzimas que lo producen, y un grupo de enzimas que lo consumen. Estas coenzimas son, por ende, continuamente creadas, consumidas y luego recicladas.[25]
La coenzima más importante es el adenosín trifosfato (ATP). Este nucleótido es usado para transferir energía química entre distintas reacciones químicas. Sólo hay una pequeña parte de ATP en las células, pero como es continuamente regenerado, el cuerpo humano puede llegar a utilizar su propio peso en ATP por día.[25] El ATP actúa como una conexión entre el catabolismo y el anabolismo, con reacciones catabólicas que generan ATP y reacciones anabólicas que lo consumen. También es útil para transportar grupos fosfato en reacciones de fosforilación.
Una vitamina es un compuesto orgánico necesitado en pequeñas cantidades que no puede ser sintetizado en las células. En la nutrición humana, la mayoría de las vitaminas trabajan como coenzimas modificadas; por ejemplo, todas las vitaminas hidrosolubles son fosforiladas o acopladas a nucleótidos cuando son utilizadas por las células.[26]
La nicotinamida adenina dinucleótido (NAD), un derivado de la vitamina B, es una importante coenzima que actúa como aceptor de protones. Cientos de deshidrogenasas eliminan electrones de sus sustratos y reducen el NAD+ en NADH. Esta forma reducida de coenzima es luego un sustrato para cualquier componente en la célula que necesite reducir su sustrato.[27] El NAD existe en dos formas relacionadas en la célula, NADH y NADPH. El NAD+/NADH es más importante en reacciones catabólicas, mientras que el NADP+/NADPH es principalmente utilizado en reacciones anabólicas.
Minerales y cofactores [editar]
Los elementos inorgánicos juegan un rol crítico en el metabolismo; algunos son abundantes (sodio y potasio, por ejemplo), mientras que otros actúan a concentraciones mínimas. Alrededor del 99% de la masa de un mamífero se encuentra compuesta por los elementos carbono, nitrógeno, calcio, sodio, cloro, potasio, hidrógeno, oxígeno y azufre.[28] Los compuestos orgánicos (proteínas, lípidos y carbohidratos) contienen, en su mayoría, carbono y nitrógeno, mientras que la mayoría del oxígeno y del hidrógeno están presentes en el agua.[28]
Los elementos inorgánicos actúan como electrolitos iónicos. Los iones de mayor importancia son sodio, potasio, calcio, magnesio, cloruro y fosfato, y el ion orgánico bicarbonato. El gradiente iónico a lo largo de las membranas de la célula mantienen la presión osmótica y el pH.[29] Los iones son también críticos para nervios y músculos ya que el potencial de acción en estos tejidos es producido por el intercambio de electrolitos entre el fluido extracelular y el citosol.[30] Los electrolitos entran y salen de la célula a través de proteínas en la membrana plasmática, denominadas canales iónicos. Por ejemplo, la contracción muscular depende del movimiento del calcio, sodio y potasio a través de los canales iónicos en la membrana y los túbulos T.[31]
Los metales de transición se encuentran presentes en el organismo principalmente como zinc y hierro, que son los más abundantes.[32] [33] Estos metales son usados en algunas proteínas como cofactores y son esenciales para la actividad de enzimas como la catalasa y proteínas transportadoras del oxígeno como la hemoglobina.[34] Estos cofactores están estrechamente ligados a una proteína; a pesar de que los cofactores de enzimas pueden ser modificados durante la catálisis, siempre tienden a volver al estado original antes de que la catálisis tuviera lugar. Los micronutrientes son captados por los organismos por medio de trasportadores específicos y proteínas de almacenamiento específicas tales como la ferritina o la metalotioneína, mientras no son utilizadas.[35] [36]
Catabolismo [editar]
El catabolismo es el conjunto de procesos metabólicos que liberan energía. Estos incluyen degradación y oxidación de moléculas de alimento, así como reacciones que retienen la energía del Sol. El propósito de estás reacciones catabólicas es proveer energía, poder reductor y componentes necesitados por reacciones anabólicas. La naturaleza de estas reacciones catabólicas difiere de organismo en organismo. Sin embargo, estas diferentes formas de catabolismo dependen de reacciones de reducción-oxidación que involucran transferencia de electrones de moléculas donantes (como las moléculas orgánicas, agua, amoníaco, sulfuro de hidrógeno e iones ferrosos), a aceptores de dichos electrones como el oxígeno, el nitrato o el sulfato.[37]
En los animales, estas reacciones conllevan la degradación de moléculas orgánicas complejas a otras más simples, como dióxido de carbono y agua. En organismos fotosintéticos como plantas y cianobacteria, estas transferencias de electrones no liberan energía, pero son usadas como un medio para almacenar energía solar.[38]
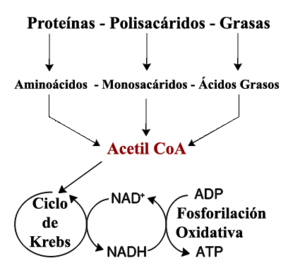
El conjunto de reacciones catabólicas más común en animales puede ser separado en tres etapas distintas. En la primera, moléculas orgánicas grandes como las proteínas, polisacáridos o lípidos son digeridos en componentes más pequeños fuera de las células. Luego, estas moléculas pequeñas son llevadas a las células y convertidas en moléculas aún más pequeñas, generalmente acetilos que se unen covalentemente a la coenzima A, para formar la acetil-coenzima A, que libera energía. Finalmente, el grupo acetil en la molécula de acetil CoA es oxidado a agua y dióxido de carbono, liberando energía que se retiene al reducir la coenzima nicotinamida adenina dinucleótido (NAD+) en NADH.
Digestión [editar]
Las macromoléculas como el almidón, la celulosa o las proteínas no pueden ser tomadas por las células automáticamente, por lo que necesitan que se degraden en unidades más simples antes de usarlas en el metabolismo celular. Muchas enzimas digieren estos polímeros. Estas enzimas incluyen peptidasa que digiere proteínas en aminoácidos, glicosil hidrolasas que digieren polisacáridos en disacáridos y monosacáridos, y lipasas que digieren los triglicéridos en ácidos grasos y glicerol.
Los microbios simplemente secretan enzimas digestivas en sus alrededores[39] [40] mientras que los animales secretan estas enzimas desde células especializadas al aparato digestivo.[41] Los aminoácidos, monosacáridos, y triglicéridos liberados por estas enzimas extracelulares son absorbidos por las células mediante proteínas específicas de transporte.[42] [43]
Energía de compuestos orgánicos [editar]
El catabolismo de carbohidratos es la degradación de los hidratos de carbono en unidades menores. Los carbohidratos son usualmente tomados por la célula una vez que fueron digeridos en monosacáridos.[44] Una vez dentro de la célula, la ruta de degradación es la glucólisis, donde los azúcares como la glucosa y la fructosa son transformados en piruvato y algunas moléculas de ATP son generadas.[45] El piruvato o ácido pirúvico es un intermediario en varias rutas metabólicas, pero la mayoría es convertido en acetil CoA y cedido al ciclo de Krebs. Aunque más ATP es generado en el ciclo, el producto más importante es el NADH, sintetizado a partir del NAD+ por la oxidación del acetil-CoA. La oxidación libera dióxido de carbono como producto de desecho. Una ruta alternativa para la degradación de la glucosa es la ruta pentosa-fosfato, que reduce la coenzima NADPH y produce azúcares de 5 carbonos como la ribosa, el azúcar que forma parte de los ácidos nucleicos.
Las grasas son catalizadas por la hidrólisis a ácidos grasos y glicerol. El glicerol entra en la glucólisis y los ácidos grasos son degradados por beta oxidación para liberar acetil CoA, que es luego cedido al nombrado ciclo de Krebs. Debido a sus proporciones altas del grupo metileno, los ácidos grasos liberan más energía en su oxidación que los carbohidratos, ya que los carbohidratos como la glucosa tienen más oxígeno en sus estructuras.
Los aminoácidos son usados principalmente para sintentizar proteínas y otras biomoléculas; sólo los excedentes son oxidados a urea y dióxido de carbono como fuente de energía.[46] Esta ruta oxidativa empieza con la eliminación del grupo amino por una aminotransferasa. El grupo amino es cedido al ciclo de la urea, dejando un esqueleto carbónico en forma de cetoácido.[47] Los aminoácidos glucogénicos pueden ser transformados en glucosa mediante gluconeogénesis.[48]
Fosforilación oxidativa [editar]

En la fosforilación oxidativa, los electrones liberados de moléculas de alimento en rutas como el ciclo de Krebs son transferidas con oxígeno, y la energía es liberada para sintetizar adenosín trifosfato. Esto se da en las células eucariotas por una serie de proteínas en las membranas de la mitocondria llamadas cadena de transporte de electrones. En las células procariotas, estas proteínas se encuentran en la membrana interna.[49] Estas proteínas utilizan la energía liberada de la oxidación del electrón que lleva la coenzima NADH para bombear protones a lo largo de la membrana.[50]
Los protones bombeados fuera de la mitocondria crean una diferencia de concentración a lo largo de la membrana, lo que genera un gradiente electroquímico.[51] Esta fuerza hace que vuelvan a la mitocondria a través de una subunidad de la ATP-sintasa. El flujo de protones hace que la subunidad menor gire, lo que produce que el sitio activo fosforile al adenosín difosfato (ADP) y lo convierta en ATP.[25]
Energía de compuestos inorgánicos [editar]
Las procariotas poseen un tipo de metabolismo donde la energía se obtiene a partir de un compuesto inorgánico. Estos organismos utilizan hidrógeno,[52] compuestos del azufre reducidos (como el sulfuro, sulfuro de hidrógeno y tiosulfato),[2] óxidos ferrosos[53] o amoníaco[54] como fuentes de poder reductor y obtienen energía de la oxidación de estos compuestos utilizando como aceptores de electrones oxígeno o nitrito.[55] Estos procesos microbióticos son importantes en ciclos biogeoquímicos como la nitrificación y la desnitrificación, esenciales para la fertilidad del suelo[56] [57]
Energía de la luz [editar]
La energía solar es captada por plantas, cianobacterias, bacterias púrpuras, bacterias verdes del azufre y algunos protistas. Este proceso está ligado a la conversión del dióxido de carbono en compuestos orgánicos, como parte de la fotosíntesis.[58] [59]
La captura de energía solar es un proceso similar en principio a la fosforilación oxidativa, ya que almacena energía en gradientes de concentración de protones, que da lugar a la síntesis de ATP.[25] Los electrones necesarios para llevar a cabo este transporte de protones provienen de una serie de proteínas denominadas centro de reacción fotosintética. Estas estructuras son clasificadas en dos dependiendo de su pigmento, siendo las bacterias quienes tienen un solo grupo, mientras que en las plantas y cianobacterias pueden ser dos.[60]
En las plantas, el fotosistema II usa energía solar para obtener los electrones del agua, liberando oxígeno como producto de desecho. Los electrones luego fluyen hacia el complejo del citocromo b6f, que usa su energía para bombear protones a lo largo de la membrana tilacoidea del cloroplasto.[38] Estos protones se mueven a través de la ATP-sintasa, mediante el mismo mecanismo explicado anteriormente. Los electrones luego fluyen por el fotosistema I y pueden ser utilizados para reducir la coenzima NADP+, que será utilizado en el ciclo de Calvin, o recicladas para la futura generación de ATP.[61]
Anabolismo [editar]
El anabolismo es el conjunto de procesos metabólicos constructivos en donde la energía liberada por el catabolismo es utilizada para sintetizar moléculas complejas. En general, las moléculas complejas que dan lugar a estructuras celulares son construidas a partir de precursores simples. El anabolismo involucra tres facetas. Primero, la producción de precursores como aminoácidos, monosacáridos, isoprenoides y nucleótidos; segundo, su activación en reactivos usando energía del ATP; y tercero, el conjunto de estos precursores en moléculas más complejas como proteínas, polisacáridos, lípidos y ácidos nucleicos.
Los organismos difieren en cuántas moléculas pueden sintetizar por sí mismos en sus células. Los organismos autótrofos, como las plantas, pueden construir moléculas orgánicas complejas y proteínas por sí mismos a partir moléculas simples como dióxido de carbono y agua. Los organismos heterótrofos, en cambio, requieren de una fuente de sustancias más complejas, como monosacáridos y aminoácidos, para producir estas moléculas complejas. Los organismos pueden ser clasificados por su fuente de energía:
- Fotoautótrofos y fotoheterótrofos, que obtienen la energía del Sol.
- Quimioheterótrofos y quimioautótrofos, que obtienen la energía mediante reacciones oxidativas.
Fijación del carbono [editar]

La fotosíntesis es la síntesis de glucosa a partir de energía solar, dióxido de carbono (CO2) y agua (H2O), con oxígeno como producto de desecho. Este proceso utiliza el ATP y el NADPH producido por los centros de reacción fotosintéticos para convertir el CO2 en 3-fosfoglicerato, que puede ser convertido en glucosa. Esta reacción de fijación del CO2 es llevada a cabo por la enzima RuBisCO como parte del ciclo de Calvin.[62] Se dan tres tipos de fotosíntesis en las plantas; fijación del carbono C3, fijación del carbono C4 y fotosíntesis CAM. Estos difieren en la vía que el CO2 sigue en el ciclo de Calvin, con plantas C3 que fijan el CO2 directamente, mientras que las fotosíntesis C4 y CAM incorporan el CO2 en otros compuestos primero como adaptaciones para soportar la luz solar intensa y las condiciones secas.[63]
En procariotas fotosintéticas, los mecanismos de la fijación son más diversos. El CO2 puede ser fijado por el ciclo de Calvin, y asimismo por el Ciclo de Krebs inverso,[64] o la carboxilación del acetil-CoA.[65] [66] Los quimioautótrofos también pueden fijar el CO2 mediante el ciclo de Calvin, pero utilizan la energía de compuestos inorgánicos para llevar a cabo la reacción.[67]
Carbohidratos [editar]
En el anabolismo de carbohidratos, se pueden sintetizar ácidos orgánicos simples desde monosacáridos como la glucosa y luego sintetizar polisacáridos como el almidón. La generación de glucosa desde compuestos como el piruvato, el ácido láctico, el glicerol y los aminoácidos es denominada gluconeogénesis. La gluconeogénesis transforma piruvato en glucosa-6-fosfato a través de una serie de intermediarios, muchos de los cuales son compartidos con la glucólisis.[45] Sin embargo, esta ruta no es simplemente la inversa a la glucólisis, ya que varias etapas son catalizadas por enzimas no glucolíticas. Esto es importante a la hora de evitar que ambas rutas estén activas a la vez dando lugar a un ciclo fútil.[68] [69]
A pesar de que la grasa es una forma común de almacenamiento de energía, en los vertebrados como los humanos, los ácidos grasos no pueden ser transformados en glucosa por gluconeogénesis, ya que estos organismos no pueden convertir acetil-CoA en piruvato.[70] Como resultado, tras un tiempo de inanición, los vertebrados necesitan producir cuerpos cetónicos desde los ácidos grasos para reemplazar la glucosa en tejidos como el cerebro, que no puede metabolizar ácidos grasos.[71] En otros organismos como las plantas y las bacterias, este problema metabólico es solucionado utilizando el ciclo del glioxilato, que sobrepasa la descarboxilación en el ciclo de Krebs y permite la transformación de acetil-CoA en ácido oxalacético, el cual puede ser utilizado en la síntesis de glucosa.[72] [70]
Los polisacáridos y los glicanos son sintetizados por medio de una adición secuencial de monosacáridos llevada a cabo por glicosil-transferasas de un donador reactivo azúcar-fosfato a un aceptor como el grupo hidroxilo en el polisacárido que se sintetiza. Como cualquiera de los grupos hidroxilos del anillo de la sustancia puede ser aceptor, los polisacáridos producidos pueden tener estructuras ramificadas o lineales.[73] Estos polisacáridos producidos pueden tener funciones metabólicas o estructurales por sí mismos o también pueden ser transferidos a lípidos y proteínas por medio de enzimas.[74] [75]
Ácidos grasos, isoprenoides y esteroides [editar]
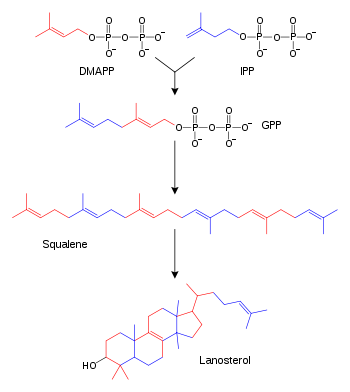
Los ácidos grasos se sintentizan al polimerizar y reducir unidades de acetil-CoA. Las cadenas en los ácidos grasos son extendidas por un ciclo de reacciones que agregan el grupo acetil, lo reducen a alcohol, deshidratan a un grupo alqueno y luego lo reducen nuevamente a un grupo alcano. Las enzimas de la síntesis de ácidos grasos se dividen en dos grupos: en los animales y hongos, las reacciones de la síntesis son llevadas a cabo por una sola proteína multifuncional tipo I,[76] mientras que en plástidos de plantas y en bacterias son las enzimas tipo II por separado las que llevan a cabo cada etapa en la ruta.[77] [78]
Los terpenos e isoprenoides son clases de lípidos que incluyen carotenoides y forman la familia más amplia de productos naturales de la planta.[79] Estos compuestos son sintentizados por la unión y modificación de unidades de isopreno donadas por los precursores reactivos pirofosfosfato isopentenil y pirofosfato dimetilalil.[80] Estos precursores pueden sintentizarse de diversos modos. En animales y archaeas, estos compuestos se sintentizan a partir de acetil-CoA,[81] mientras que en plantas y bacterias se hace a partir de piruvato y gliceraldehído 3-fosfato como sustratos.[82] [80] Una reacción que usa estos donadores isoprénicos activados es la biosíntesis de esteroides. En este caso, las unidades de isoprenoides son unidas covalentemente para formar escualeno, que se pliega formando una serie de anillos dando lugar a una molécula denominada lanosterol.[83] El lanosterol puede luego ser transformado en esteroides como el colesterol.
Proteínas [editar]
La habilidad de los organismos para sintetizar los 20 aminoácidos conocidos varía. Las bacterias y las plantas pueden sintetizar los 20, pero los mamíferos pueden sintetizar solo los diez aminoácido no esenciales.[17] Por ende, los aminoácidos esenciales deben ser obtenidos del alimento. Todos los aminoácidos son sintetizados por intermediarios en la glucólisis y el ciclo de Krebs. El nitrógeno es obtenido por el ácido glutámico y la glutamina. La síntesis de aminoácidos depende en la formación apropiada del ácido alfa-keto, que luego es transaminado para formar un aminoácido.[84]
Los aminoácidos son sintetizados en proteínas al ser unidos en una cadena por enlaces peptídicos. Cada proteína diferente tiene una secuencia única e irrepetible de aminoácidos: esto es la estructura primaria. Los aminoácidos pueden formar una gran variedad de proteínas dependiendo de la secuencia de estos en la proteína. Las proteínas son constituidas por aminoácidos que han sido activados por la adición de un ARNt a través de un enlace éster.[85] El aminoacil-ARNt es entonces un sustrato para el ribosoma, que va añadiendo los residuos de aminoácidos a la cadena proteica, sobre la base de la secuencia de información que va "leyendo" el ribosoma en una molécula de ARN mensajero.[86]
Síntesis de nucleótidos [editar]
Los nucleótidos son sintetizados a partir de aminoácidos, dióxido de carbono y ácido fórmico en rutas que requieren una cantidad mayor de energía metabólica.[87] [88] En consecuencia, la mayoría de los organismos tienen un sistema eficiente para resguardar los nucleótidos preformados.[87] [89] Las purinas son sintetizadas como nucleósidos (bases unidas a ribosa). Tanto la adenina como la guanina son sintetizadas a partir de un precursor nucleósido, la inosina monofosfato, que es sintetizada usando átomos de los aminoácidos glicina, glutamina y ácido aspártico; también ocurre lo mismo con el HCOO− que es transferido desde la coenzima tetrahidrofolato. Las pirimidinas, en cambio, son sintetizadas desde el ácido orótico, que a su vez es sintetizado a partir de la glutamina y el aspartato.[90]
Xenobióticos y metabolismo reductor [editar]
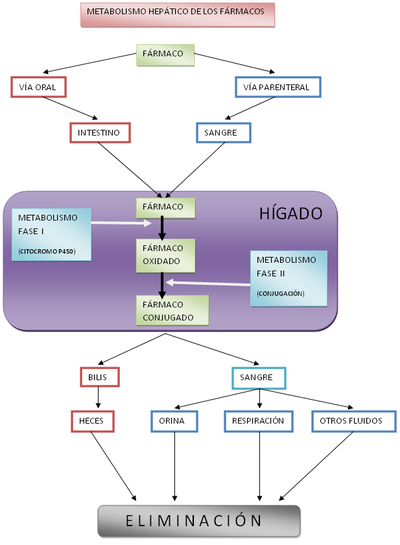
Todos los organismos se encuentran constantemente expuestos a compuestos y elementos químicos que no pueden utilizar como alimento y serían dañinos si se acumularan en sus células, ya que no tendrían una función metabólica. Estos compuestos potencialmente dañinos son llamados xenobióticos.[91] Los xenobióticos como las drogas sintéticas, los venenos naturales y los antibióticos son detoxificados por un conjunto de enzimas xenobióticas-metabolizadoras. En los humanos, esto incluye a las citocromo oxidasas P450,[92] las UDP-glucuroniltransferasas[93] y las glutation-S-transferasas.[94]
Este sistema de enzimas actúa en tres etapas. En primer lugar, oxida los xenobióticos (fase I) y luego conjuga grupos solubles al agua en la molécula (fase II). El xenobiótico modificado puede ser extraído de la célula por exocitosis y, en organismos pluricelulares, puede ser más metabolizado antes de ser excretado (fase III). En ecología, estas reacciones son particularmente importantes por la biodegradación microbiana de agentes contaminantes y la biorremediación de tierras contaminadas.[95] Muchas de estas reacciones microbióticas son compartidas con organismos pluricelulares, pero debido a la mayor biodiversidad de microbios, éstos son capaces de tratar con un rango más amplio de xenobióticos en contraste a los que pueden llevar a cabo los organismos pluricelulares; los microbios pueden incluso degradar agentes contaminantes como compuestos organoclorados.[96]
Un problema relacionado con los organismos aeróbicos es el estrés oxidativo.[97] Sin embargo, una bacteria estresada podría ser más efectiva para la degradación de estos contaminantes.[98]
Los procesos como la fosforilación oxidativa y la formación de enlaces disulfuro durante el plegamiento de proteínas producen especies reactivas del oxígeno como el peróxido de hidrógeno.[99] Estos oxidantes dañinos son neutralizados por metabolitos antioxidantes como el glutation y por enzimas como las catalasas y las peroxidasas.[100] [101]
Un ejemplo de metabolismo xenobiótico es la depuración de los fármacos por parte del hígado, como puede verse en el diagrama adjunto.
Homeostasis: regulación y control [editar]
Debido a que el ambiente de los organismos cambia constantemente, las reacciones metabólicas son reguladas para mantener un conjunto de condiciones en la célula, una condición denominada homeostasis.[102] [103] Esta regulación permite a los organismos responder a estímulos e interactuar con el ambiente.[104] Para entender cómo son controladas las vías metabólicas, existen dos conceptos vinculados. En primer lugar, la regulación de una enzima en una ruta es cómo incrementa o disminuye su actividad en respuesta a señales o estímulos. En segundo lugar, el control llevado a cabo por esta enzima viene dado por los efectos que, dichos cambios de su actividad, tienen sobre la velocidad de la ruta (el flujo de la ruta).[105] Por ejemplo, una enzima muestra cambios en su actividad; pero si estos cambios tienen un efecto mínimo en el flujo de la ruta metabólica, entonces esta enzima no se relaciona con el control de la ruta.[106]
Existen múltiples niveles para regular el metabolismo. En la regulación intrínseca, la ruta metabólica se autorregula para responder a cambios en los niveles de sustratos o productos; por ejemplo, una disminución en la cantidad de productos puede incrementar el flujo en la ruta para compensarlo.[105] Este tipo de regulación suele implicar una regulación alostérica de las actividades de las distintas enzimas en la ruta.[107] El control extrínseco implica a una célula en un organismo pluricelurar, cambiando su metabolismo en respuesta a señales de otras células. Estas señales son enviadas generalmente en forma de mensajeros como las hormonas, y los factores de crecimiento, que son detectados por receptores celulares específicos en la superficie de la célula.[108] Estas señales son transmitidas hacia el interior de la célula mediante mensajeros secundarios que generalmente involucran la fosforilación de proteínas.[109]
Un ejemplo de control extrínseco es la regulación del metabolismo de la glucosa mediante la hormona denominada insulina.[110] La insulina es producida como consecuencia de un aumento de la concentración de azúcar en la sangre. La unión de esta hormona a los receptores de insulina activa una cascada de proteín-quinasas que estimulan la absorción de glucosa por parte de la célula para transformarla en moléculas de almacenamiento como los ácidos grasos y el glucógeno.[111] El metabolismo del glucógeno es controlado por la actividad de la glucógeno fosforilasa, enzima que degrada el glucógeno, y la glucógeno sintasa, enzima que lo sintetiza. Estas enzimas son reguladas de un modo recíproco, siendo la fosforilación la que inhibe a la glucógeno sintentasa, pero activando a su vez a la glucógeno fosforilasa. La insulina induce la síntesis de glucógeno al activar fosfatasas y producir una disminución en la fosforilación de estas enzimas.[112]
Termodinámica de los organismos vivos [editar]
Los organismos vivos deben respetar las leyes de la termodinámica. La segunda ley de la termodinámica establece que en cualquier sistema cerrado, la cantidad de entropía tendrá una tendencia a incrementar. A pesar de que la complejidad de los organismos vivos contradice esta ley, la vida es posible ya que todos los organismos vivos son sistemas abiertos que intercambian materia y energía con sus alrededores. Por ende, los sistemas vivos no se encuentran en equilibrio, sino que son sistemas de disipación que mantienen su estado de complejidad ya que provocan incrementos mayores en la entropía de sus alrededores.[113] El metabolismo de una célula logra esto mediante la relación entre los procesos espontáneos del catabolismo con los procesos no-espontáneos del anabolismo. En términos termodinámicos, el metabolismo mantiene el orden al crear un desorden.[114]
Véase también [editar]
- Respiración celular
- Síntesis de proteínas
- Calorimetría
- Metabolismo microbiano
- Clasificación nutricional básica
Referencias [editar]
- ↑ «Metabolismo». Enciclopedia Médica. MedlinePlus (2006-10-25). Consultado el 26-10-2007.
- ↑ a b Friedrich C (1998) «Physiology and genetics of sulfur-oxidizing bacteria» Adv Microb Physiol. Vol. 39. pp. 235-89. PMID 9328649.
- ↑ Smith E, Morowitz H (2004) «Universality in intermediary metabolism» Proc Natl Acad Sci U S A. Vol. 101. n.º 36. pp. 13168-73. PMID 15340153.
- ↑ Ebenhöh O, Heinrich R (2001) «Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems» Bull Math Biol. Vol. 63. n.º 1. pp. 21-55. PMID 11146883.
- ↑ Meléndez-Hevia E, Waddell T, Cascante M (1996) «The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution» J Mol Evol. Vol. 43. n.º 3. pp. 293-303. PMID 8703096.
- ↑ Rennie M (1999) «An introduction to the use of tracers in nutrition and metabolism» Proc Nutr Soc. Vol. 58. n.º 4. pp. 935-44. PMID 10817161.
- ↑ Phair R (1997) «Development of kinetic models in the nonlinear world of molecular cell biology» Metabolism. Vol. 46. n.º 12. pp. 1489-95. PMID 9439549.
- ↑ Sterck L, Rombauts S, Vandepoele K, Rouzé P, Van de Peer Y (2007) «How many genes are there in plants (... and why are they there)?» Curr Opin Plant Biol. Vol. 10. n.º 2. pp. 199-203. PMID 17289424.
- ↑ Borodina I, Nielsen J (2005) «From genomes to in silico cells via metabolic networks» Curr Opin Biotechnol. Vol. 16. n.º 3. pp. 350-5. PMID 15961036.
- ↑ Gianchandani E, Brautigan D, Papin J (2006) «Systems analyses characterize integrated functions of biochemical networks» Trends Biochem Sci. Vol. 31. n.º 5. pp. 284-91. PMID 16616498.
- ↑ Thykaer J, Nielsen J (2003) «Metabolic engineering of beta-lactam production» Metab Eng. Vol. 5. n.º 1. pp. 56-69. PMID 12749845..
- ↑ González-Pajuelo M, Meynial-Salles I, Mendes F, Andrade J, Vasconcelos I, Soucaille P «Metabolic engineering of Clostridium acetobutylicum for the industrial production of 1,3-propanediol from glycerol» Metab Eng. Vol. 7. n.º 5-6. pp. 329-36. PMID 16095939.
- ↑ Krämer M, Bongaerts J, Bovenberg R, Kremer S, Müller U, Orf S, Wubbolts M, Raeven L (2003) «Metabolic engineering for microbial production of shikimic acid» Metab Eng. Vol. 5. n.º 4. pp. 277-83. PMID 14642355.
- ↑ Koffas M, Roberge C, Lee K, Stephanopoulos G (1999) «Metabolic engineering» Annu Rev Biomed Eng. Vol. 1. pp. 535-57. PMID 11701499.
- ↑ Michie K, Löwe J (2006) «Dynamic filaments of the bacterial cytoskeleton» Annu Rev Biochem. Vol. 75. pp. 467-92. PMID 16756499.
- ↑ «La célula, estructura y fisiología» (PDF). Consejería de Educación. Gobierno de Canarias. Consultado el 26-10-2007.
- ↑ a b c d e Nelson DL, Cox MM (2005). Lehninger Principles of Biochemistry. New York: W. H. Freeman and company, pp. 841. ISBN 0-7167-4339-6.
- ↑ Fahy E, Subramaniam S, Brown H, Glass C, Merrill A, Murphy R, Raetz C, Russell D, Seyama Y, Shaw W, Shimizu T, Spener F, van Meer G, VanNieuwenhze M, White S, Witztum J, Dennis E (2005) «A comprehensive classification system for lipids» J Lipid Res. Vol. 46. n.º 5. pp. 839-61. PMID 15722563.
- ↑ «Nomenclature of Lipids». IUPAC-IUB Commission on Biochemical Nomenclature (CBN). Consultado el 08-03-2007.
- ↑ Hegardt F (1999) «Mitochondrial 3-hydroxy-3-methylglutaryl-CoA synthase: a control enzyme in ketogenesis» Biochem J. Vol. 338 (Pt 3). pp. 569-82. PMID 10051425.
- ↑ Raman R, Raguram S, Venkataraman G, Paulson J, Sasisekharan R (2005) «Glycomics: an integrated systems approach to structure-function relationships of glycans» Nat Methods. Vol. 2. n.º 11. pp. 817-24. PMID 16278650.
- ↑ Sierra S, Kupfer B, Kaiser R (2005) «Basics of the virology of HIV-1 and its replication» J Clin Virol. Vol. 34. n.º 4. pp. 233-44. PMID 16198625.
- ↑ a b Wimmer M, Rose I (1978) «Mechanisms of enzyme-catalyzed group transfer reactions» Annu Rev Biochem. Vol. 47. pp. 1031-78. PMID 354490.
- ↑ Mitchell P (1979) «The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems» Eur J Biochem. Vol. 95. n.º 1. pp. 1-20. PMID 378655.
- ↑ a b c d Dimroth P, von Ballmoos C, Meier T (2006) «Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series» EMBO Rep. Vol. 7. n.º 3. pp. 276-82. PMID 16607397.
- ↑ Ann Coulton, John Kerner, Joane Hattner y Ashini Srivastava (2006) «Stanford School of Medicine Nutrition Courses»
- ↑ Pollak N, Dölle C, Ziegler M (2007) «The power to reduce: pyridine nucleotides--small molecules with a multitude of functions» Biochem J. Vol. 402. n.º 2. pp. 205-18. PMID 17295611.
- ↑ a b Heymsfield S, Waki M, Kehayias J, Lichtman S, Dilmanian F, Kamen Y, Wang J, Pierson R (1991) «Chemical and elemental analysis of humans in vivo using improved body composition models» Am J Physiol. Vol. 261. n.º 2 Pt 1. pp. E190-8. PMID 1872381.
- ↑ Sychrová H (2004) «Yeast as a model organism to study transport and homeostasis of alkali metal cations» Physiol Res. Vol. 53 Suppl 1. pp. S91-8. PMID 15119939.
- ↑ Levitan I (1988) «Modulation of ion channels in neurons and other cells» Annu Rev Neurosci. Vol. 11. pp. 119-36. PMID 2452594.
- ↑ Dulhunty A (2006) «Excitation-contraction coupling from the 1950s into the new millennium» Clin Exp Pharmacol Physiol. Vol. 33. n.º 9. pp. 763-72. PMID 16922804.
- ↑ Mahan D, Shields R (1998) «Macro- and micromineral composition of pigs from birth to 145 kilograms of body weight» J Anim Sci. Vol. 76. n.º 2. pp. 506-12. PMID 9498359.
- ↑ Husted S, Mikkelsen B, Jensen J, Nielsen N (2004) «Elemental fingerprint analysis of barley (Hordeum vulgare) using inductively coupled plasma mass spectrometry, isotope-ratio mass spectrometry, and multivariate statistics» Anal Bioanal Chem. Vol. 378. n.º 1. pp. 171-82. PMID 14551660.
- ↑ Finney L, O'Halloran T (2003) «Transition metal speciation in the cell: insights from the chemistry of metal ion receptors» Science. Vol. 300. n.º 5621. pp. 931-6. PMID 12738850.
- ↑ Cousins R, Liuzzi J, Lichten L (2006) «Mammalian zinc transport, trafficking, and signals» J Biol Chem. Vol. 281. n.º 34. pp. 24085-9. PMID 16793761.
- ↑ Dunn L, Rahmanto Y, Richardson D (2007) «Iron uptake and metabolism in the new millennium» Trends Cell Biol. Vol. 17. n.º 2. pp. 93-100. PMID 17194590.
- ↑ Nealson K, Conrad P (1999) «Life: past, present and future» Philos Trans R Soc Lond B Biol Sci. Vol. 354. n.º 1392. pp. 1923-39. PMID 10670014.
- ↑ a b Nelson N, Ben-Shem A (2004) «The complex architecture of oxygenic photosynthesis» Nat Rev Mol Cell Biol. Vol. 5. n.º 12. pp. 971-82. PMID 15573135.
- ↑ Häse C, Finkelstein R (1993) «Bacterial extracellular zinc-containing metalloproteases» Microbiol Rev. Vol. 57. n.º 4. pp. 823-37. PMID 8302217.
- ↑ Gupta R, Gupta N, Rathi P (2004) «Bacterial lipases: an overview of production, purification and biochemical properties» Appl Microbiol Biotechnol. Vol. 64. n.º 6. pp. 763-81. PMID 14966663.
- ↑ Hoyle T (1997) «The digestive system: linking theory and practice» Br J Nurs. Vol. 6. n.º 22. pp. 1285-91. PMID 9470654.
- ↑ Souba W, Pacitti A (1992) «How amino acids get into cells: mechanisms, models, menus, and mediators» JPEN J Parenter Enteral Nutr. Vol. 16. n.º 6. pp. 569-78. PMID 1494216.
- ↑ Barrett M, Walmsley A, Gould G (1999) «Structure and function of facilitative sugar transporters» Curr Opin Cell Biol. Vol. 11. n.º 4. pp. 496-502. PMID 10449337.
- ↑ Bell G, Burant C, Takeda J, Gould G (1993) «Structure and function of mammalian facilitative sugar transporters» J Biol Chem. Vol. 268. n.º 26. pp. 19161-4. PMID 8366068.
- ↑ a b Bouché C, Serdy S, Kahn C, Goldfine A (2004) «The cellular fate of glucose and its relevance in type 2 diabetes» Endocr Rev. Vol. 25. n.º 5. pp. 807-30. PMID 15466941.
- ↑ Sakami W, Harrington H (1963) «Amino acid metabolism» Annu Rev Biochem. Vol. 32. pp. 355-98. PMID 14144484.
- ↑ Brosnan J (2000) «Glutamate, at the interface between amino acid and carbohydrate metabolism» J Nutr. Vol. 130. n.º 4S Suppl. pp. 988S-90S. PMID 10736367.
- ↑ Young V, Ajami A (2001) «Glutamine: the emperor or his clothes?» J Nutr. Vol. 131. n.º 9 Suppl. pp. 2449S-59S; discussion 2486S-7S. PMID 11533293.
- ↑ Hosler J, Ferguson-Miller S, Mills D (2006) «Energy transduction: proton transfer through the respiratory complexes» Annu Rev Biochem. Vol. 75. pp. 165-87. PMID 16756489.
- ↑ Schultz B, Chan S (2001) «Structures and proton-pumping strategies of mitochondrial respiratory enzymes» Annu Rev Biophys Biomol Struct. Vol. 30. pp. 23-65. PMID 11340051.
- ↑ Capaldi R, Aggeler R (2002) «Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor» Trends Biochem Sci. Vol. 27. n.º 3. pp. 154-60. PMID 11893513.
- ↑ Friedrich B, Schwartz E (1993) «Molecular biology of hydrogen utilization in aerobic chemolithotrophs» Annu Rev Microbiol. Vol. 47. pp. 351-83. PMID 8257102.
- ↑ Weber K, Achenbach L, Coates J (2006) «Microorganisms pumping iron: anaerobic microbial iron oxidation and reduction» Nat Rev Microbiol. Vol. 4. n.º 10. pp. 752-64. PMID 16980937.
- ↑ Jetten M, Strous M, van de Pas-Schoonen K, Schalk J, van Dongen U, van de Graaf A, Logemann S, Muyzer G, van Loosdrecht M, Kuenen J (1998) «The anaerobic oxidation of ammonium» FEMS Microbiol Rev. Vol. 22. n.º 5. pp. 421-37. PMID 9990725.
- ↑ Simon J (2002) «Enzymology and bioenergetics of respiratory nitrite ammonification» FEMS Microbiol Rev. Vol. 26. n.º 3. pp. 285-309. PMID 12165429.
- ↑ Conrad R (1996) «Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O, and NO)» Microbiol Rev. Vol. 60. n.º 4. pp. 609-40. PMID 8987358.
- ↑ Barea J, Pozo M, Azcón R, Azcón-Aguilar C (2005) «Microbial co-operation in the rhizosphere» J Exp Bot. Vol. 56. n.º 417. pp. 1761-78. PMID 15911555.
- ↑ van der Meer M, Schouten S, Bateson M, Nübel U, Wieland A, Kühl M, de Leeuw J, Sinninghe Damsté J, Ward D (2005) «Diel variations in carbon metabolism by green nonsulfur-like bacteria in alkaline siliceous hot spring microbial mats from Yellowstone National Park» Appl Environ Microbiol. Vol. 71. n.º 7. pp. 3978-86. PMID 16000812.
- ↑ Tichi M, Tabita F (2001) «Interactive control of Rhodobacter capsulatus redox-balancing systems during phototrophic metabolism» J Bacteriol. Vol. 183. n.º 21. pp. 6344-54. PMID 11591679.
- ↑ Allen J, Williams J (1998) «Photosynthetic reaction centers» FEBS Lett. Vol. 438. n.º 1-2. pp. 5-9. PMID 9821949.
- ↑ Munekage Y, Hashimoto M, Miyake C, Tomizawa K, Endo T, Tasaka M, Shikanai T (2004) «Cyclic electron flow around photosystem I is essential for photosynthesis» Nature. Vol. 429. n.º 6991. pp. 579-82. PMID 15175756.
- ↑ Miziorko H, Lorimer G (1983) «Ribulose-1,5-bisphosphate carboxylase-oxygenase» Annu Rev Biochem. Vol. 52. pp. 507-35. PMID 6351728.
- ↑ Dodd A, Borland A, Haslam R, Griffiths H, Maxwell K (2002) «Crassulacean acid metabolism: plastic, fantastic» J Exp Bot. Vol. 53. n.º 369. pp. 569-80. PMID 11886877.
- ↑ Hügler M, Wirsen C, Fuchs G, Taylor C, Sievert S (2005) «Evidence for autotrophic CO2 fixation via the reductive tricarboxylic acid cycle by members of the epsilon subdivision of proteobacteria» J Bacteriol. Vol. 187. n.º 9. pp. 3020-7. PMID 15838028.
- ↑ Strauss G, Fuchs G (1993) «Enzymes of a novel autotrophic CO2 fixation pathway in the phototrophic bacterium Chloroflexus aurantiacus, the 3-hydroxypropionate cycle» Eur J Biochem. Vol. 215. n.º 3. pp. 633-43. PMID 8354269.
- ↑ Wood H (1991) «Life with CO or CO2 and H2 as a source of carbon and energy» FASEB J. Vol. 5. n.º 2. pp. 156-63. PMID 1900793.
- ↑ Shively J, van Keulen G, Meijer W (1998) «Something from almost nothing: carbon dioxide fixation in chemoautotrophs» Annu Rev Microbiol. Vol. 52. pp. 191-230. PMID 9891798.
- ↑ Boiteux A, Hess B (1981) «Design of glycolysis» Philos Trans R Soc Lond B Biol Sci. Vol. 293. n.º 1063. pp. 5-22. PMID 6115423.
- ↑ Pilkis S, el-Maghrabi M, Claus T (1990) «Fructose-2,6-bisphosphate in control of hepatic gluconeogenesis. From metabolites to molecular genetics» Diabetes Care. Vol. 13. n.º 6. pp. 582-99. PMID 2162755.
- ↑ a b Ensign S (2006) «Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation» Mol Microbiol. Vol. 61. n.º 2. pp. 274-6. PMID 16856935.
- ↑ Finn P, Dice J «Proteolytic and lipolytic responses to starvation» Nutrition. Vol. 22. n.º 7-8. pp. 830-44. PMID 16815497.
- ↑ Kornberg H, Krebs H (1957) «Synthesis of cell constituents from C2-units by a modified tricarboxylic acid cycle» Nature. Vol. 179. n.º 4568. pp. 988-91. PMID 13430766.
- ↑ Rademacher T, Parekh R, Dwek R (1988) «Glycobiology» Annu Rev Biochem. Vol. 57. pp. 785-838. PMID 3052290.
- ↑ Opdenakker G, Rudd P, Ponting C, Dwek R (1993) «Concepts and principles of glycobiology» FASEB J. Vol. 7. n.º 14. pp. 1330-7. PMID 8224606.
- ↑ McConville M, Menon A (2000) «Recent developments in the cell biology and biochemistry of glycosylphosphatidylinositol lipids (review)» Mol Membr Biol. Vol. 17. n.º 1. pp. 1-16. PMID 10824734.
- ↑ Chirala S, Wakil S (2004) «Structure and function of animal fatty acid synthase» Lipids. Vol. 39. n.º 11. pp. 1045-53. PMID 15726818.
- ↑ White S, Zheng J, Zhang Y (2005) «The structural biology of type II fatty acid biosynthesis» Annu Rev Biochem. Vol. 74. pp. 791-831. PMID 15952903.
- ↑ Ohlrogge J, Jaworski J (1997) «Regulation of fatty acid synthesis» Annu Rev Plant Physiol Plant Mol Biol. Vol. 48. pp. 109-136. PMID 15012259.
- ↑ Dubey V, Bhalla R, Luthra R (2003) «An overview of the non-mevalonate pathway for terpenoid biosynthesis in plants» J Biosci. Vol. 28. n.º 5. pp. 637-46. PMID 14517367.
- ↑ a b Kuzuyama T, Seto H (2003) «Diversity of the biosynthesis of the isoprene units» Nat Prod Rep. Vol. 20. n.º 2. pp. 171-83. PMID 12735695.
- ↑ Grochowski L, Xu H, White R (2006) «Methanocaldococcus jannaschii uses a modified mevalonate pathway for biosynthesis of isopentenyl diphosphate» J Bacteriol. Vol. 188. n.º 9. pp. 3192-8. PMID 16621811.
- ↑ Lichtenthaler H (1999) «The 1-Ddeoxy-D-xylulose-5-phosphate pathway of isoprenoid biosynthesis in plants» Annu Rev Plant Physiol Plant Mol Biol. Vol. 50. pp. 47-65. PMID 15012203.
- ↑ Schroepfer G (1981) «Sterol biosynthesis» Annu Rev Biochem. Vol. 50. pp. 585-621. PMID 7023367.
- ↑ Guyton, Arthur C.; John E. Hall (2006). Textbook of Medical Physiology. Philadelphia: Elsevier, pp. 855-6. ISBN 0-7216-0240-1.
- ↑ Ibba M, Söll D (2001) «The renaissance of aminoacyl-tRNA synthesis» EMBO Rep. Vol. 2. pp. 382-7. PMID 11375928.
- ↑ Lengyel P, Söll D (1969) «Mechanism of protein biosynthesis» Bacteriol Rev. Vol. 33. n.º 2. pp. 264-301. PMID 4896351.
- ↑ a b Rudolph F (1994) «The biochemistry and physiology of nucleotides» J Nutr. Vol. 124. n.º 1 Suppl. pp. 124S-127S. PMID 8283301.
- ↑ Zrenner R, Stitt M, Sonnewald U, Boldt R (2006) «Pyrimidine and purine biosynthesis and degradation in plants» Annu Rev Plant Biol. Vol. 57. pp. 805-36. PMID 16669783.
- ↑ Stasolla C, Katahira R, Thorpe T, Ashihara H (2003) «Purine and pyrimidine nucleotide metabolism in higher plants» J Plant Physiol. Vol. 160. n.º 11. pp. 1271-95. PMID 14658380.
- ↑ Smith J (1995) «Enzymes of nucleotide synthesis» Curr Opin Struct Biol. Vol. 5. n.º 6. pp. 752-7. PMID 8749362.
- ↑ Testa B, Krämer S (2006) «The biochemistry of drug metabolism--an introduction: part 1. Principles and overview» Chem Biodivers. Vol. 3. n.º 10. pp. 1053-101. PMID 17193224.
- ↑ Danielson P (2002) «The cytochrome P450 superfamily: biochemistry, evolution and drug metabolism in humans» Curr Drug Metab. Vol. 3. n.º 6. pp. 561-97. PMID 12369887.
- ↑ King C, Rios G, Green M, Tephly T (2000) «UDP-glucuronosyltransferases» Curr Drug Metab. Vol. 1. n.º 2. pp. 143-61. PMID 11465080.
- ↑ Sheehan D, Meade G, Foley V, Dowd C (2001) «Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily» Biochem J. Vol. 360. n.º Pt 1. pp. 1-16. PMID 11695986.
- ↑ Galvão T, Mohn W, de Lorenzo V (2005) «Exploring the microbial biodegradation and biotransformation gene pool» Trends Biotechnol. Vol. 23. n.º 10. pp. 497-506. PMID 16125262.
- ↑ Janssen D, Dinkla I, Poelarends G, Terpstra P (2005) «Bacterial degradation of xenobiotic compounds: evolution and distribution of novel enzyme activities» Environ Microbiol. Vol. 7. n.º 12. pp. 1868-82. PMID 16309386.
- ↑ Davies K (1995) «Oxidative stress: the paradox of aerobic life» Biochem Soc Symp. Vol. 61. pp. 1-31. PMID 8660387.
- ↑ Una bacteria "estresada" puede ser más eficiente - La Nación, 24 de mayo de 2006.
- ↑ Tu B, Weissman J (2004) «Oxidative protein folding in eukaryotes: mechanisms and consequences» J Cell Biol. Vol. 164. n.º 3. pp. 341-6. PMID 14757749.
- ↑ Sies H (1997) «Oxidative stress: oxidants and antioxidants» Exp Physiol. Vol. 82. n.º 2. pp. 291-5. PMID 9129943.
- ↑ Vertuani S, Angusti A, Manfredini S (2004) «The antioxidants and pro-antioxidants network: an overview» Curr Pharm Des. Vol. 10. n.º 14. pp. 1677-94. PMID 15134565.
- ↑ Albert R (2005) «Scale-free networks in cell biology» J Cell Sci. Vol. 118. n.º Pt 21. pp. 4947-57. PMID 16254242.
- ↑ Brand M (1997) «Regulation analysis of energy metabolism» J Exp Biol. Vol. 200. n.º Pt 2. pp. 193-202. PMID 9050227.
- ↑ Soyer O, Salathé M, Bonhoeffer S (2006) «Signal transduction networks: topology, response and biochemical processes» J Theor Biol. Vol. 238. n.º 2. pp. 416-25. PMID 16045939.
- ↑ a b Salter M, Knowles R, Pogson C (1994) «Metabolic control» Essays Biochem. Vol. 28. pp. 1-12. PMID 7925313.
- ↑ Westerhoff H, Groen A, Wanders R (1984) «Modern theories of metabolic control and their applications (review)» Biosci Rep. Vol. 4. n.º 1. pp. 1-22. PMID 6365197.
- ↑ Fell D, Thomas S (1995) «Physiological control of metabolic flux: the requirement for multisite modulation» Biochem J. Vol. 311 (Pt 1). pp. 35-9. PMID 7575476.
- ↑ Hendrickson W (2005) «Transduction of biochemical signals across cell membranes» Q Rev Biophys. Vol. 38. n.º 4. pp. 321-30. PMID 16600054.
- ↑ Cohen P (2000) «The regulation of protein function by multisite phosphorylation--a 25 year update» Trends Biochem Sci. Vol. 25. n.º 12. pp. 596-601. PMID 11116185.
- ↑ Lienhard G, Slot J, James D, Mueckler M (1992) «How cells absorb glucose» Sci Am. Vol. 266. n.º 1. pp. 86-91. PMID 1734513.
- ↑ Roach P (2002) «Glycogen and its metabolism» Curr Mol Med. Vol. 2. n.º 2. pp. 101-20. PMID 11949930.
- ↑ Newgard C, Brady M, O'Doherty R, Saltiel A (2000) «Organizing glucose disposal: emerging roles of the glycogen targeting subunits of protein phosphatase-1» Diabetes. Vol. 49. n.º 12. pp. 1967-77. PMID 11117996.
- ↑ von Stockar U, Liu J (1999) «Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth» Biochim Biophys Acta. Vol. 1412. n.º 3. pp. 191-211. PMID 10482783.
- ↑ Demirel Y, Sandler S (2002) «Thermodynamics and bioenergetics» Biophys Chem. Vol. 97. n.º 2-3. pp. 87-111. PMID 12050002.
Bibliografía [editar]
Introductoria
- Rose S, Mileusnic R. The Chemistry of Life. (Penguin Press Science, 1999), ISBN 0-14-027273-9
- Schneider ED, Sagan D. Into the Cool: Energy Flow, Thermodynamics, and Life. (University Of Chicago Press, 2005), ISBN 0-226-73936-8
- Lane N. Oxygen: The Molecule that Made the World. (Oxford University Press, USA, 2004), ISBN 0-19-860783-0
Avanzada
- Price N, Stevens L. Fundamentals of Enzymology: Cell and Molecular Biology of Catalytic Proteins. (Oxford University Press, 1999), ISBN 0-19-850229-X
- Berg J, Tymoczko J, Stryer L. Biochemistry. (W. H. Freeman and Company, 2002), ISBN 0-7167-4955-6
- Cox M, Nelson DL. Lehninger Principles of Biochemistry. (Palgrave Macmillan, 2004), ISBN 0-7167-4339-6
- Brock TD, Madigan MT, Martinko J, Parker J. Brock's Biology of Microorganisms. (Benjamin Cummings, 2002), ISBN 0-13-066271-2
- Da Silva JJRF, Williams RJP. The Biological Chemistry of the Elements: The Inorganic Chemistry of Life. (Clarendon Press, 1991), ISBN 0-19-855598-9
- Nicholls DG, Ferguson SJ. Bioenergetics. (Academic Press Inc., 2002), ISBN 0-12-518121-3
Enlaces externos [editar]
 Wikcionario tiene definiciones para metabolismo.
Wikcionario tiene definiciones para metabolismo.
En Español [editar]
Información general
- En MedlinePlus puede encontrar más información sobre Metabolismo
- Monografías de metabolismo
Glosarios
Metabolismo humano
En Inglés [editar]
General Information (Información General)
- Interactive Flow Chart of the Major Metabolic Pathways – Tabla interactiva de las rutas metabólicas mayores
- Metabolism, Cellular Respiration and Photosynthesis – Metabolismo, respiración celular y fotosíntesis
- The Biochemistry of Metabolism – La Bioquímica del Metabolismo
Human metabolism (Metabolismo humano)
- Topics in Medical Biochemistry – Temas en la Bioquímica Medicinal
- The Medical Biochemistry Page – La Página de Medicina Bioquímica
0 comentarios